Neue Veröffentlichung in "Communications Biology"
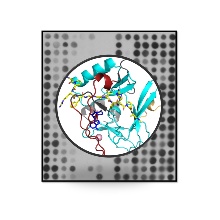
Die Methylierung von Lysin Resten in Proteinen ist eine häufig vorkommende post-translationale Proteinmodifikation mit wichtigen biologischen Funktionen. Als Ausgangpunkt dieser Arbeit haben wir die Spezifität von SETD2 systematisch analysiert. SETD2 ist eine menschlichen Protein-Lysin-Methyltransferase mit starken Krankheitsverbindungen, die Lysin 36 in Histon H3 (H3K36) methyliert. Unter Verwendung eines Peptidarray-basierten Ansatzes haben wir das Spezifitätsprofil von SETD2 bestimmt und neue Nicht-Histon-Substrate dieses Enzyms identifiziert. Bemerkenswerterweise beobachteten wir, dass SETD2 an mehreren Stellen andere Aminosäuren als die in der natürlichen H3K36-Sequenz vorhandenen bevorzugt, und auf dieser Grundlage konnten wir ein SETD2-Supersubstrat entwerfen, das etwa 290-fach stärker methyliert war als H3K36, das derzeit beste bekanntes SETD2-Substrat. Die sehr starke Methylierung des Supersubstrats wurde auf Peptidarrays mit gereinigten Peptiden, Proteinen in vitro und auch in Zellen beobachtet. Wir haben die Struktur von SETD2 gelöst, das an das Supersubstrat gebunden ist, und so verbesserte molekulare Wechselwirkungen als möglichen Grund für die erhöhte Methylierung des Supersubstrats sowie Konformationsänderungen identifiziert, die neue Einblicke in den Mechanismus von SETD2 liefern. Unsere Daten zeigen, dass das Substratsequenzdesign die Aktivität von Protein-Lysin-Methyltransferasen stark erhöhen kann.